Importando Moléculas por Nombre
Seleccione el menú "Archivo".
Luego seleccione "Importar" y "Buscar por nombre químico ..."
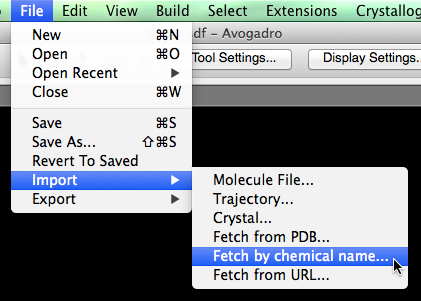
Aparecerá un cuadro de diálogo (que se muestra a continuación), donde puede escribir cualquier nombre químico.
Avogadro importará la molécula en la pantalla de visualización después de hacer clic en "Aceptar".
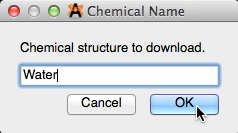
La descarga de la molécula en línea puede demorar unos segundos o incluso un minuto. Avogadro utiliza el "Resolvente químico" de los NIH http://cactus.nci.nih.gov/chemical/structurepara convertir el nombre en una estructura molecular.
mportación desde el banco de datos de proteínas (PDB)
Puede leer los archivos de PDB que descargue usted mismo desde http://www.rcsb.org/ o acceder al código de PDB usted mismo.
Importando directamente
Las versiones más antiguas de Avogadro tienen un error con el acceso directo al PDB (ya que el sitio web se ha movido), pero con v 1.2.0, puede usar de nuevo Archivo> Importar> Recuperar desde PDB ... para descargar proteínas.
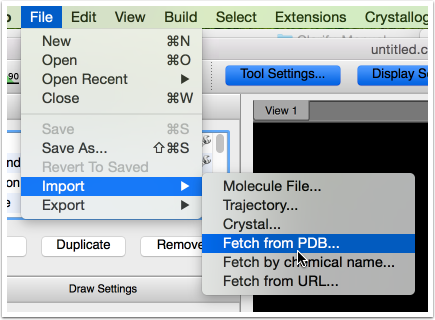
Aparecerá un cuadro de diálogo que le permitirá ingresar un código PDB (por ejemplo, 1CRN para crambin)
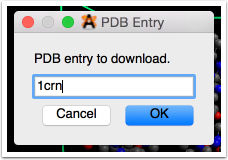
Ahí está ... los datos del PDB directamente desde el sitio web.
Leyendo un archivo PDB descargado
Si la importación directa no funciona, también puede utilizar el sitio web usted mismo. Vaya a http://www.rcsb.org/ y seleccione una proteína, luego descargue el formato “Archivo PDB (texto)” del archivo. Desde la carpeta de descargas, puede arrastrar y soltar el archivo en una pantalla de Avogadro, o arrastrar y soltar el archivo sobre el ícono de la aplicación Avogadro.
Al arrastrar y soltar el archivo se abrirá la importación de PDB con una celda de unidad (Avogadro muestra las celdas de unidad para todas las importaciones de PDB).
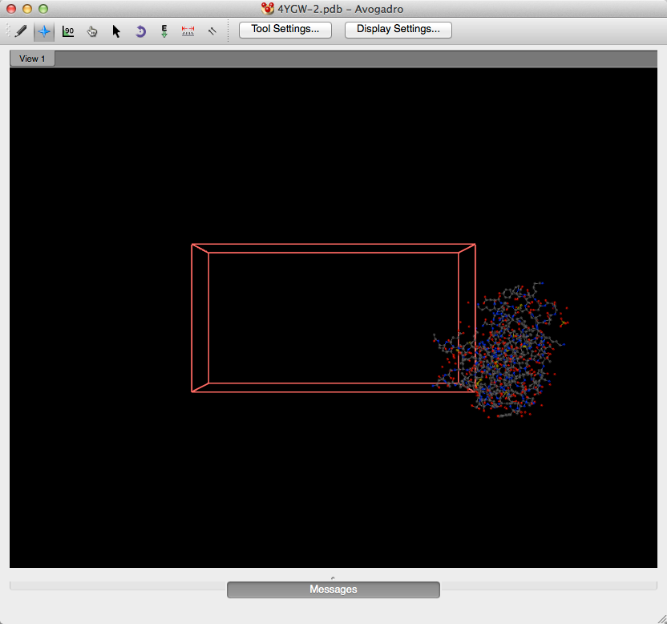
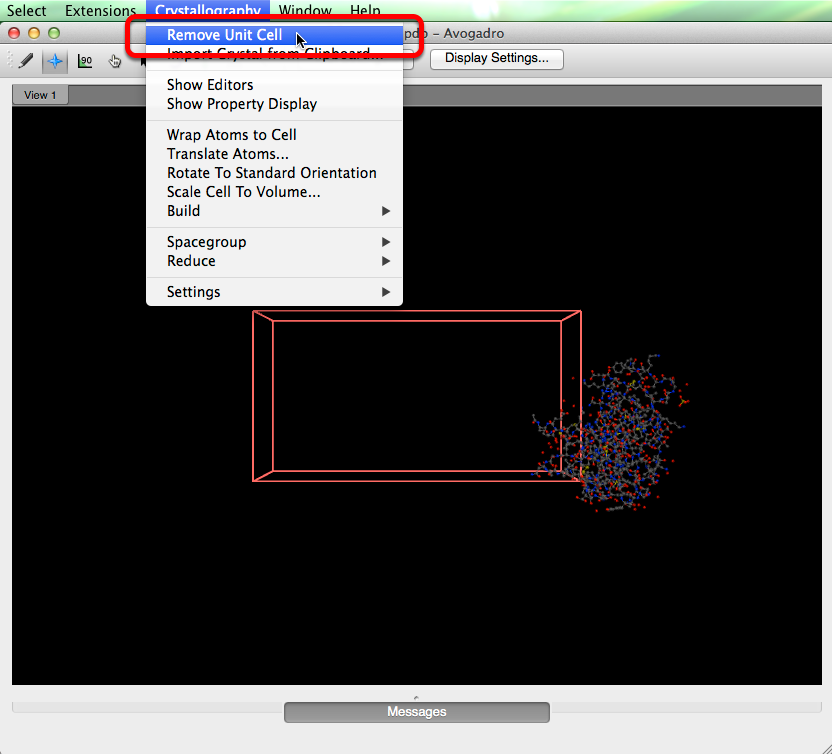
Si la celda de la unidad no es deseada, puede eliminar la celda de la unidad en el menú "Cristalografía" seleccionando "Eliminar celda de la unidad".
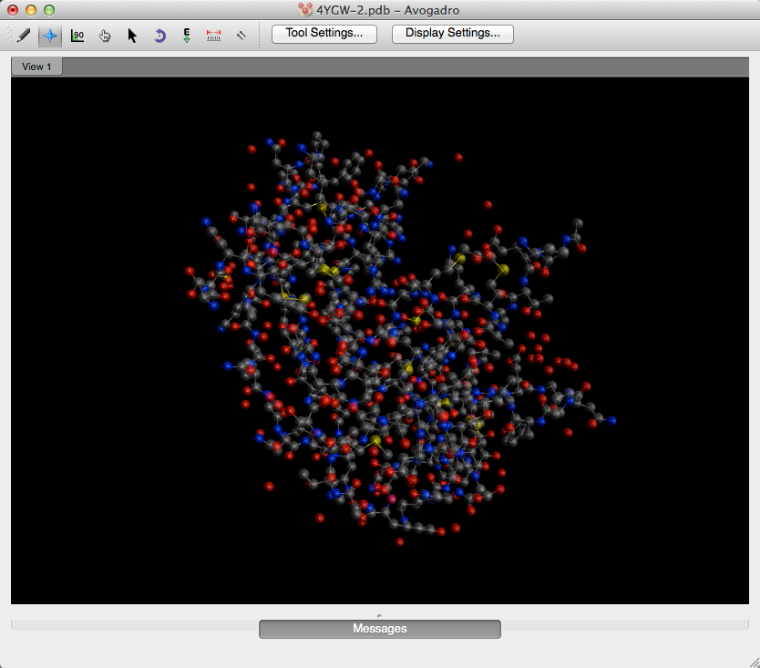
Construyendo un péptido
Un tutorial sobre cómo crear un modelo de péptidos personalizado en Avogadro.
Seleccione el menú "Construir".
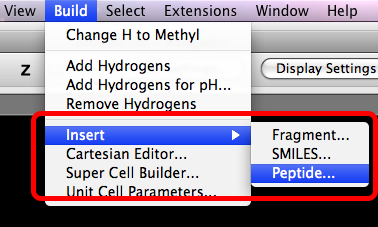
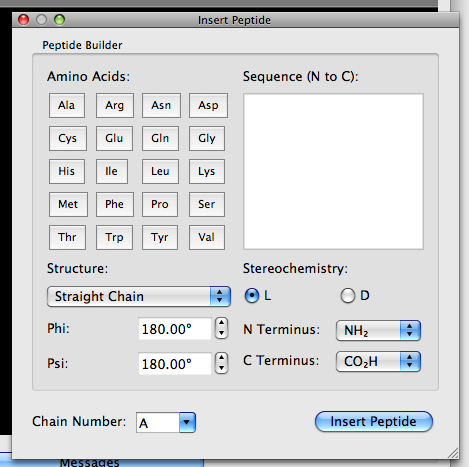
Abre la ventana del generador de péptidos. Puede seleccionar aminoácidos para insertar en el nuevo péptido.
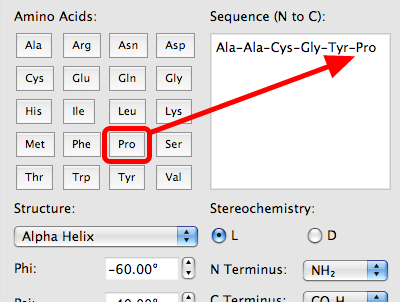
Al hacer clic en determinados aminoácidos, se agregarán a la secuencia de la derecha. El péptido se acumulará como una secuencia, a partir del extremo N. Por supuesto, también puede escribir los residuos directamente o pegarlos desde una base de datos en línea.
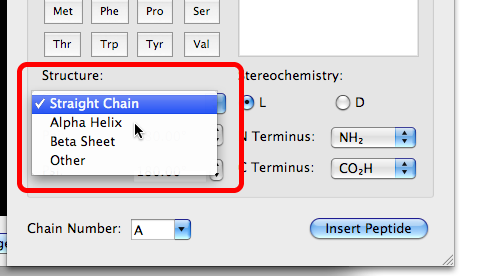
Puedes elegir la estructura secundaria.
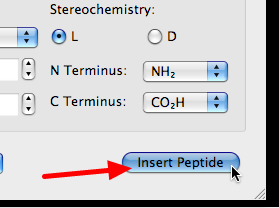
Haga clic para insertar la secuencia en la ventana principal. El nuevo oligopéptido se seleccionará automáticamente, y la herramienta de manipulación le permitirá traducir y rotar la cadena a la posición que desee.
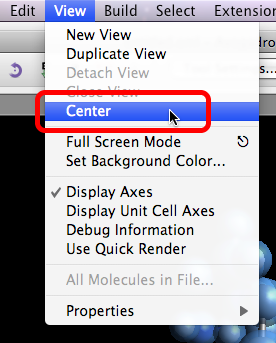
Es posible que desee volver a centrar la vista, ya que el nuevo péptido puede ser grande.
Construyendo ADN / ARN
Avogadro ahora tiene un constructor para secuencias de ácidos nucleicos y este recorrido le mostrará cómo usarlo.
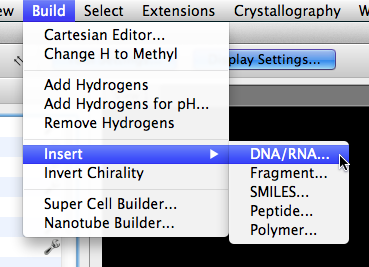
El generador de ADN / ARN se encuentra en el menú "Crear" y en el submenú "Insertar".
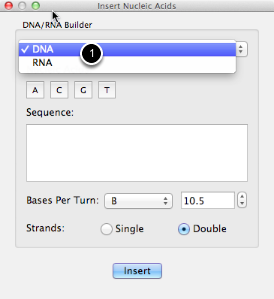
Seleccione ADN o ARN (1) y el resto de la ventana se actualizará en consecuencia.
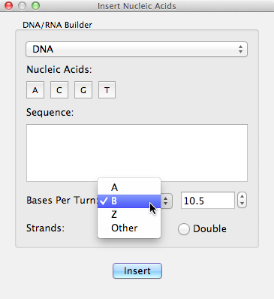
También puede controlar el número de bases por turno como se muestra (con valores predeterminados para A-DNA, B-DNA, Z-DNA o RNA).
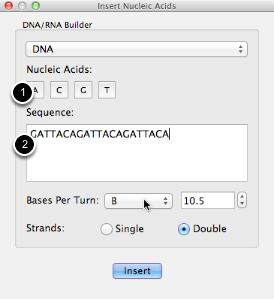
Puede ingresar la secuencia haciendo clic en los botones o escribiendo los códigos de una letra directamente. Para las secuencias de ADN (como se muestra aquí), puede insertar ADN de cadena simple o de cadena doble.
Es posible que desee volver a centrar la vista o alinearla con los ejes para ver toda la molécula.

¡Ahí vamos, la conocida doble hélice del ADN!
Construcción de nanotubos de carbono
Avogadro 1.1 incluye un nuevo constructor de nanotubos, basado en el conocido código TubeGen y el sitio web del grupo Doren en U. Delaware. ( http://turin.nss.udel.edu/research/tubegenonline.html )
Bajo el menú Construir, hay una nueva opción para el generador de nanotubos. Por el momento, solo los nanotubos de pared simple (SWNT) se pueden construir en un solo paso, aunque es fácil generar varios tubos anidados para paredes múltiples (MWNT) como se muestra aquí.
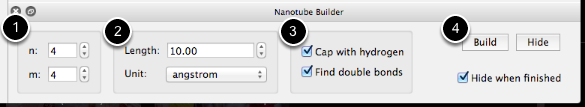
El constructor se mostrará en la parte inferior de la ventana de Avogadro. Puede configurar los índices n, m para determinar el tipo de nanotubos (1) la longitud del tubo (2), en Angstrom, bohr, picómetros, nanómetros o celdas unitarias periódicas (por ejemplo, si desea hacer un cálculo con condiciones limitadas periódicas), y cómo terminar el nanotubo (3). NOTA : determinar los dobles enlaces puede llevar mucho tiempo en los nanotubos grandes.

El nanotubo se generará alineado a lo largo del eje z, por lo que es posible que desee volver a centrar la vista.
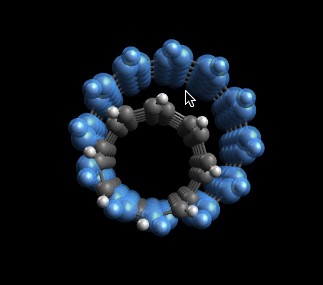
Aquí, hemos agregado un nanotubo 6,6 después de insertar nuestro nanotubo 4,4. Tendremos que volver a centrar el tubo para producir un sistema de doble pared más preciso.
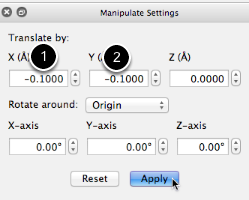
Aquí, usamos las opciones de traducción manual, nuevas en Avogadro 1.1, para "empujar" el nanotubo 6,6 en el plano XY para centrarse adecuadamente alrededor del nanotubo 4,4.
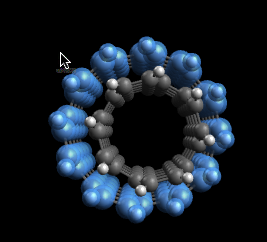
Aquí hemos empujado el tubo 6,6 a una posición aproximadamente correcta. Ahora usaremos los campos de fuerza integrados de Avogadro y la herramienta de optimización automática para relajar la estructura.
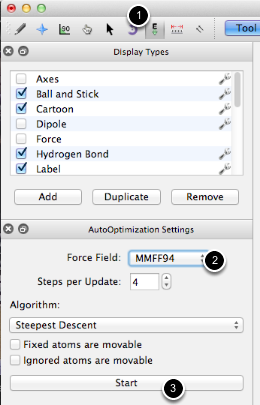
Nosotros (1) seleccionamos la herramienta Auto-Optimize para permitir la minimización interactiva de los nanotubos, y (2) seleccionamos el campo de fuerza MMFF94. Otros campos de fuerza también podrían funcionar bien. Finalmente (3) se inicia la optimización.

Después de unos pocos pasos, puede ver un nanotubo de doble pared muy relajado. Podrías repetir el proceso como desees.
Insertar Fragmentos
Avogadro incluye más de 300 moléculas comunes y fragmentos moleculares para facilitar la construcción de estructuras más grandes.
Bajo el menú "Crear", mantenga el cursor sobre "Insertar" y luego seleccione "Fragmento ...".
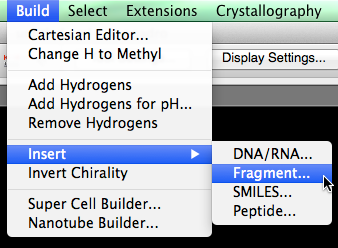
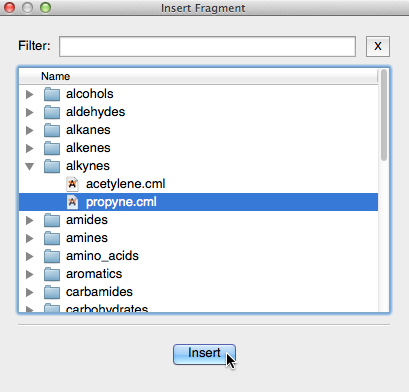
Luego aparecerá una base de datos de fragmentos (se muestra a continuación). Puede filtrar la selección si es necesario.
Después de haber hecho su selección de fragmentos, haga clic en "Insertar".
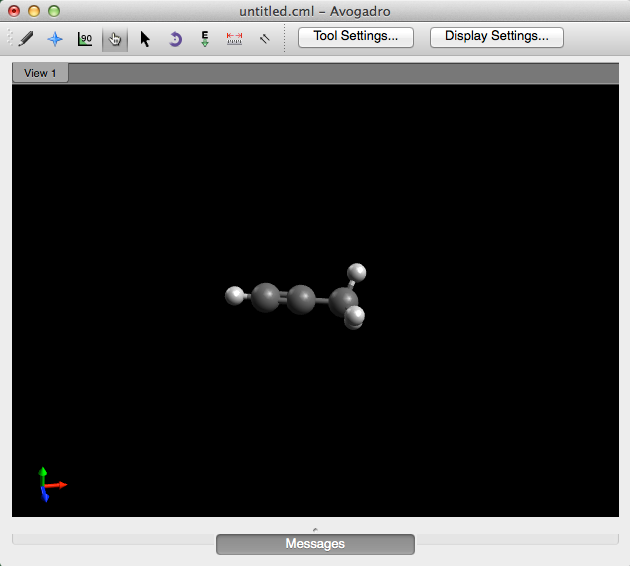
El fragmento se insertará y se seleccionará la herramienta Manipular para que pueda mover el nuevo fragmento alrededor de la ventana.
Edificio con SMILES
SMILES (sistema de entrada de línea de entrada molecular simplificada) le permite construir moléculas a través de una cadena de texto. Si tiene una cadena SMILES (p. Ej., Copiada de un papel o sitio web) o prefiere ingresar una para una molécula complicada, Avogadro creará una geometría 3D a partir de SMILES.
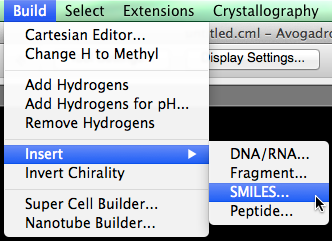
Debajo del menú "Crear", mantenga el cursor sobre "Insertar" y seleccione "SMILES ...".
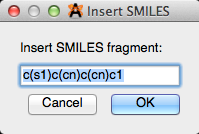
Ingrese su fragmento de SMILES, y seleccione "OK".
Ahí está..
No hay comentarios:
Publicar un comentario